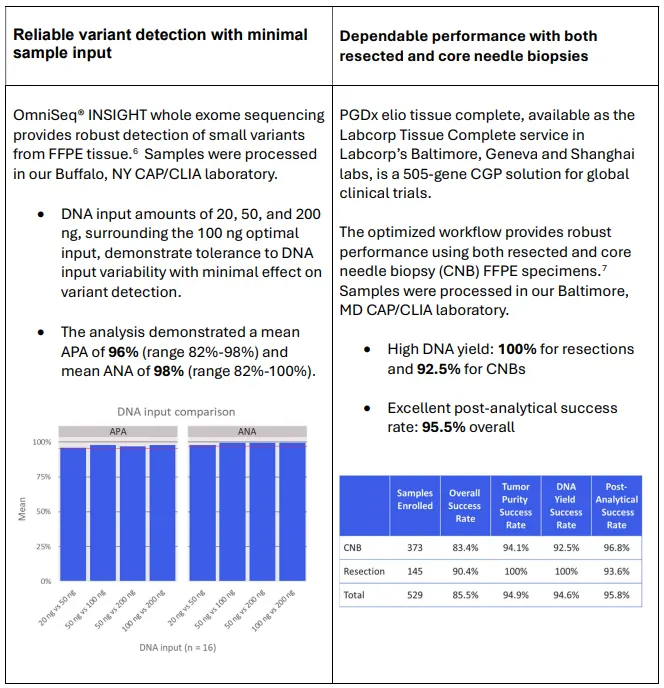
Biomarker-testen is van vitaal belang voor oncologisch onderzoek en klinische zorg. Het stelt ons in staat om gerichte therapieën te ontwikkelen en gepersonaliseerde behandelingen aan te bieden. Met behulp van next-generation sequencing (NGS) kunnen we diverse genomische veranderingen in een enkel assay detecteren. Echter, de brede toepassing hiervan wordt belemmerd door de kwaliteit en hoeveelheid beschikbare tumormonsters. Het probleem van monsterkwaliteit Een blijvend probleem bij biomarker-testen is de afhankelijkheid van formalinefixeerde, in paraffin ingesloten (FFPE) weefselmonsters. FFPE is de standaard voor monstersconservering, maar het extraheren van hoogwaardig DNA en RNA voor NGS is moeilijk. Veel klinische monsters, vooral naaldbiopsieën, leveren vaak slechts enkele nanogrammen bruikbaar materiaal op. Dr. Erin Newburn, PhD, Directeur van Field Applications bij Labcorp, benadrukt het wijdverspreide karakter van dit probleem: “Klinische proefmonsters uit FFPE-blokken hebben vaak een lage nucleïnezuuropbrengst en degradatie. Dit beïnvloedt de extractie, bibliotheekpreparatie en post-sequencing dataverwerking.” Waarde ontsluiten met geoptimaliseerde workflows en schaalbare technologieën Om deze beperkingen het hoofd te bieden, nemen laboratoria standaardwerkprocessen over die het maximale datagebruik uit moeilijke monsters maximaliseren. Labcorp heeft geautomatiseerde dubbele extractieprotocollen ontwikkeld om de nucleïnezuuropbrengst en kwaliteit uit FFPE-weefsels te verbeteren, waardoor het aantal monsters met onvoldoende hoeveelheid (QNS) wordt verminderd. “Geautomatiseerde, gestandaardiseerde workflows verbeteren de nucleïnezuuropbrengst en -kwaliteit significant”, zegt Dr. Newburn. “Dit maakt succesvolle genomische profilering en uiteindelijk betere patiëntuitkomsten mogelijk.” Naast extractie helpen kwaliteitscontrole (QC) metrieken om betrouwbare variantoproepen te garanderen. QC-tools filteren laagbetrouwbare gegevens en minimaliseren vals-positieve resultaten, waardoor betrouwbare gegevens worden geleverd om op basis daarvan informatieve beslissingen te nemen. Brede genomische oplossingen voor maximale inzichten Technologieën zoals whole-exome sequencing (WES), whole-transcriptome sequencing (WTS) en grote gerichte panelen zijn een sleutelstrategie om variantdetectie te maximaliseren uit minimale input. Voegt Dr. Newburn toe: “Comprehensieve platforms helpen biopharma-teams om data-outputs voor huidige en toekomstige biomarkers van belang te maximaliseren uit beperkt materiaal.” Beste praktijken voor succes Om uitgebreide genomische profilering (CGP) in settings met beperkte monsters te optimaliseren, is vroege afstemming met ervaren genomische aanbieders van cruciaal belang. Beste praktijken omvatten: Gestandaardiseerde extractie voor reproduceerbaarheid en schaalbaarheid QC-geïnformeerde bibliotheekpreparatie om de gegevenskwaliteit te verbeteren en herwerkzaamheid te verminderen Geavanceerde sequencingplatforms die inzichten leveren uit monsters met lage input Deze benaderingen verbeteren uitkomsten, versnellen tijdlijnen en verminderen kosten – cruciaal in klinische en onderzoeksomgevingen. Casusstudies: Het mogelijk maken van hoogbetrouwbare variantdetectie voor uw biomarker-gebaseerde proef Toestemming verleend door Labcorp Op het moment dat CGP integraal wordt voor oncologische ontwikkeling, is het genereren van betrouwbare gegevens uit beperkte monsters essentieel. Door innovaties in extractie, sequencing en analyse helpt Labcorp om het volledige potentieel van precieze oncologie te ontsluiten, één monster per keer. Partner met Labcorp om uw oncologieproeven te versnellen met schaalbare, precieze oplossingen. Bronnen 1 Hedegaard J, Thorsen K, Lund MK, et al. PLoS One. 2014;9(5):e98187. doi:10.1371/journal.pone.0098187 2 Spencer DH, Sehn JK, Abel HJ, Watson MA, Pfeifer JD, Duncavage EJ. J Mol Diagn. 2013;15(5):623-633. doi:10.1016/j.jmoldx.2013.05.004 3 McDonough SJ, Bhagwate A, Sun Z, et al. PLoS One. 2019;14(4):e0211400. doi:10.1371/journal.pone.0211400 4 Amirault K, Collins M, Beker L, et al. SLAS Technol. 2025;31:100252. doi:10.1016/j.slast.2025.100252 5 Do H, Dobrovic A. Clin Chem. 2015;61(1):64-71. doi:10.1373/clinchem.2014.223040 6 An J, Collins MJ, DeBlasi EA, et al. Gepubliceerd 23 november 2024 https://oncology.labcorp.com/analytical-validation-omniseqr-insight-whole-exome-sequencing-assay-facilitate-precision-oncology 7 Gegevens op bestand, Labcorp Gecategoriseerd onder: Klinische proeven